南湖新闻网讯(通讯员 黄臆丞)1月5日,作物遗传改良全国重点实验室张建伟教授团队联合美国堪萨斯州立大学、亚利桑那大学等单位,在Genome Biology在线发表题为“Genetic diversity and architectural dynamics of soybean centromeres”的研究论文。该研究基于三个大豆(WM82,ZH13,Jack)端粒到端粒(T2T)高质量基因组序列,绘制出大豆着丝粒卫星重复序列的全景图谱,首次系统解析了两种核心着丝粒重复序列的遗传多样性与结构动态,提出了一种全新的着丝粒DNA复制模型,为植物着丝粒功能研究、基因挖掘和基因组演化解析提供了新的理论框架。
着丝粒是动粒形成的基础结构,动粒通过与纺锤体微管结合,确保染色体在细胞分裂过程中被精确分离。尽管着丝粒在功能上高度保守,但其DNA序列却表现出显著的快速演化和高度多样性,这一现象被称为“着丝粒悖论”。在多数真核生物中,着丝粒区域富集大量卫星重复序列,但这些重复序列与着丝粒特异性组蛋白CENH3(动物中对应CENP-A)之间的功能关系仍不清楚。此前研究表明,大豆着丝粒主要由两类串联卫星重复序列构成:CentGm-1(约91 bp)和CentGm-4(约410 bp)。不同类型串联重复序列如何形成、是否承担不同功能,以及在快速演化过程中如何维持着丝粒稳定性,成为亟待解答的科学问题。
本研究发现,大豆着丝粒中的CentGm-1和CentGm-4并非随机分布,而是呈“簇状交替”的方式排列,同时着丝粒区域富含Gypsy类LTR反转录转座子(TE)。这些TE倾向插入在CentGm簇边界及变异高的区域,可能在连接或打断卫星重复阵列中发挥调控作用。研究进一步表明,高相似性且缺乏TE插入的CentGm簇,是导致不同大豆着丝粒卫星重复序列长度和组成差异的关键因素(图1)。
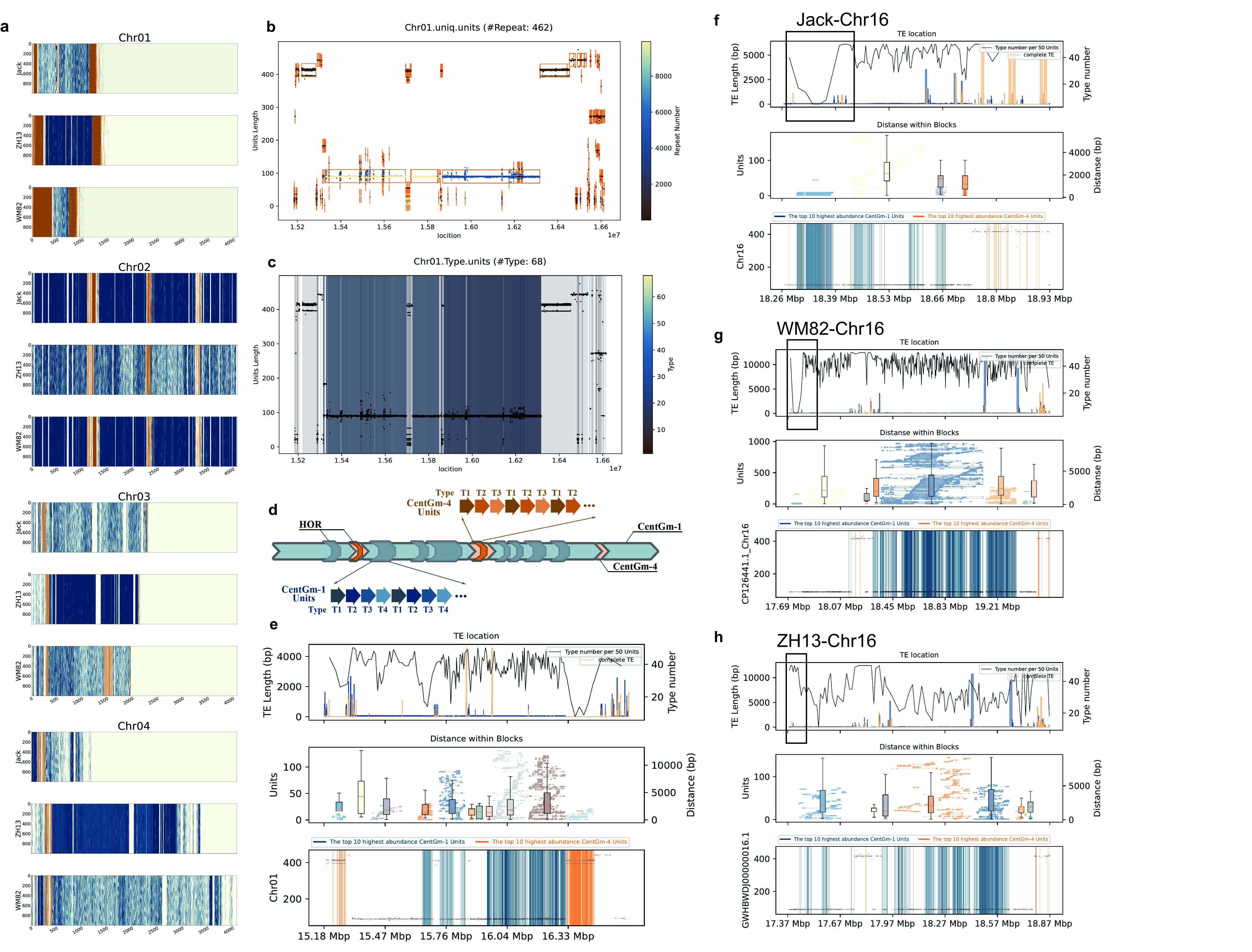
图1. 大豆两种着丝粒卫星重复序列(CentGm-1和CentGm-4)
为探究着丝粒重复序列长度变异与演化机制,研究团队在每条染色体上筛选出高频出现的CentGm-1和CentGm-4单体(HF-CentGm)。这些高频单体在着丝粒区域呈交替排列,形成不含TE插入的高阶重复阵列(HOR)。系统发育分析显示,尽管CentGm-1与CentGm-4在同一染色体上物理距离很近,但HF-CentGm呈现出截然不同的聚类模式(图2),表明这两类卫星重复很可能经历了彼此独立的演化历程。值得注意的是,同一HOR内相似单体的间距相似高度一致,结合短基因组片段难以发生高频重组的事实,暗示着丝粒可能存在特殊的复制机制。
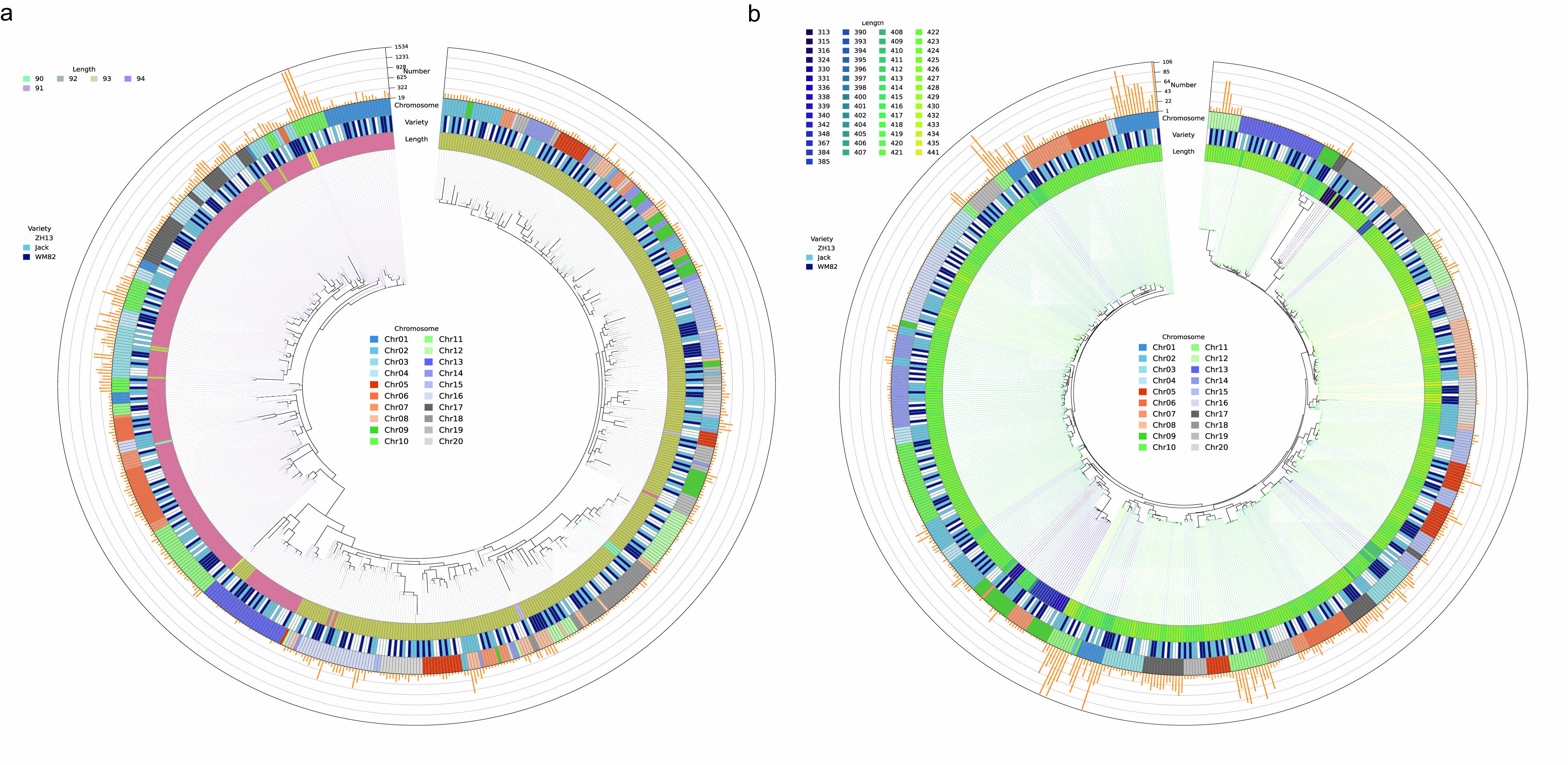
图2. HF-CentGm的系统发育分析
进一步分析三个大豆基因组中所有CentGm单体后,研究发现CentGm-1广泛分布于多条染色体,而CentGm-4则表现出染色体特异性,并在不同品种间保守。这一结果暗示CentGm-4可能与染色体特异性识别相关,而CentGm-1更可能参与着丝粒的高效复制。统计分析显示,不同大豆品种中相同类型的CentGm-1或CentGm-4单体间距无显著差异,而两种类型单体之间的间距差异极为显著(图3)。基于此,作者提出,大规模CentGm阵列可能起源于少量HF-CentGm单体类型,经多轮突变与重组逐步扩增,进一步支持了两类卫星重复序列独立起源和独立演化的假说。
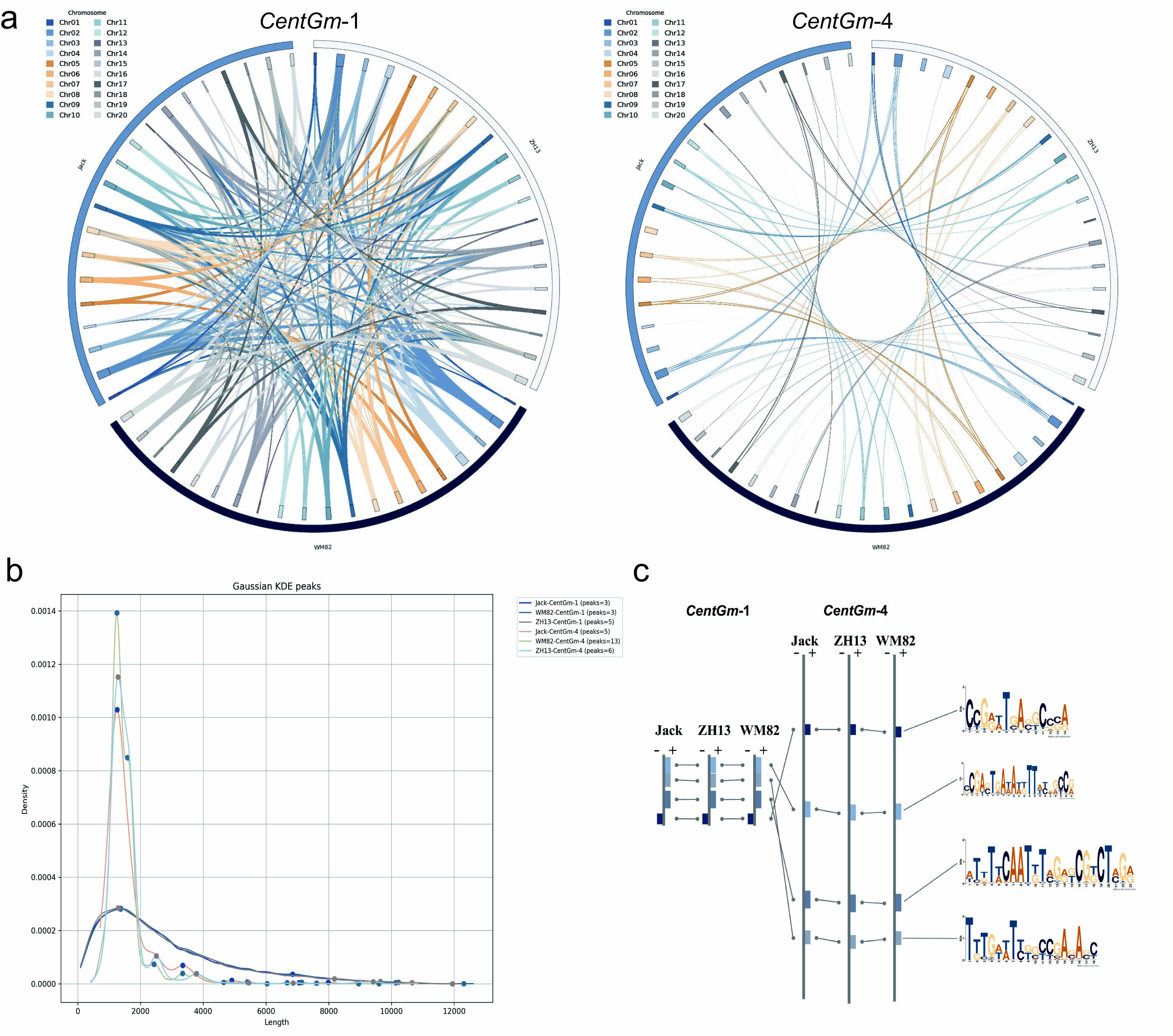
图3. 不同大豆品种CentGm单体的分布与特征分析
在此基础上,研究首次在植物中鉴定出4个全新的类“CEN-B box”的潜在CENH3结合基序。结合着丝粒卫星重复序列的空间分布和单体距离特征,作者提出了一种全新的着丝粒DNA复制模型:CentGm保守基序与CENH3结合形成复制模板,通过类似端粒酶的模板依赖型聚合酶机制以及染色体外环状DNA(eccDNA)扩增,促进HOR阵列的形成和扩张,驱动重复序列富集;TE插入则在CentGm阵列交界处引入变异,最终导致不同品种间CentGm簇长度差异,即着丝粒结构的差异(图4)。间期细胞的FISH实验中检测到多个独立的CentGm信号,为该“多起点、模板依赖”的复制假说提供了实验支持。
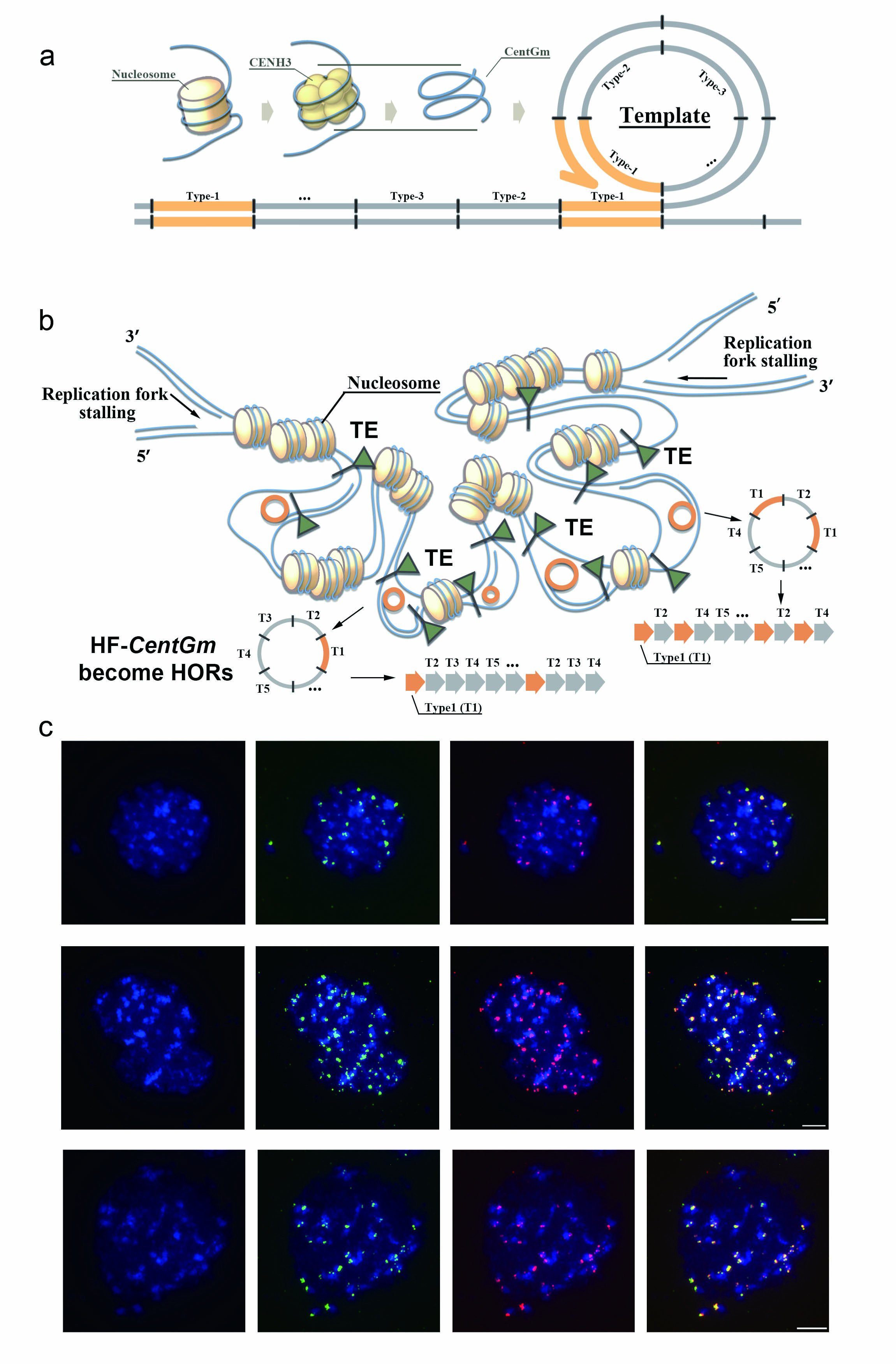
图4. 着丝粒重复区域的复制假说模型
综上,该研究系统解析了大豆基因组中复杂的着丝粒区域,深入阐明了两类主要CentGm卫星重复序列的组织特征和演化规律,首次在植物中推定潜在的CENH3结合基序,并提出了创新性的着丝粒DNA复制模型。这些发现为理解着丝粒的多样性与动态演化机制提供了重要新视角,也为未来着丝粒功能探索和关键基因挖掘奠定了理论基础。
本研究由华中农业大学作物遗传改良全国重点实验室/湖北洪山实验室牵头完成,并联合美国堪萨斯州立大学、亚利桑那大学等单位共同开展。华中农业大学黄臆丞博士(现为中国农业科学院农业基因组研究所博士后)为论文第一作者,张建伟教授为通讯作者。陈春丽教授、苏汉东教授,以及博士生官恩来、宋世鹏参与了相关研究工作。华中农业大学作物遗传改良全国重点实验室和信息技术中心超算平台为本研究提供了重要的生物信息学计算支持。该研究得到了农业生物育种国家科技重大专项、湖北洪山实验室重大项目等项目资助。
论文链接:https://doi.org/10.1186/s13059-025-03924-9
审核人:张建伟
南湖新闻网链接:https://news.hzau.edu.cn/info/1010/60478.htm